به درس دوم: “ناحیه بندی مفهومی تصاویر X-Ray ریه با پایتون” خوش آمدید.
در این درس ما از روش U-Net برای تقسیم بندی یا ناحیه بندی مفهومی نواحی ریه از تصاویر اشعه ایکس که از قفسه سینه گرفته می شود استفاد می کنیم.
در اینجا از ابزار MD.ai annotator برای دیدن تصاویر DICOM پزشکی استفاده می کنیم. همچنین از کتابخانه کلاینت پایتون MD.ai استفاده می کنیم تا تصاویر پزشکی رو دانلود کنیم و با استفاده از اون دیتاستمون رو آماده کنیم که برای آموزش مدلمون برای کلاسبندی استفاده میشه.
خب! برای شروع وارد محسط google colab بشید و مراحل زیر را به ترتیب انجام بدید:
قدم اول: اضافه کردن کتابخانه mdai:
با دستور زیر کتابخانه mdai رو نصب کنید (مثال اینجا ورژن نصبی ما ۰.۴.۱ است.)
%%bash
pip install -q -U mdai
import mdai
mdai.__version__
'۰.۴.۱'
قدم دوم: ایجاد بستر دریافت اطلاعات از mdai: یا همون mdai client:
برای نصب کلاینت mdai ما نیاز به یک توکن دسترسی (access token) داریم تا به عنوان یک کاربر در سرور mdai شناخته بشیم. برای ایجاد یک توکن یا دسترسی به توکن از قبل دریافت شده باید به سایت MD.ai بریم و یک حساب کاربری بسازیم و در صفحه تنظیمات (settings page) به بخش Personal Access Tokens بریم و توکنمونو دریافت کنیم. (توجه داشته باشید که نباید توکن خودتونو به کسی بدید)
mdai_client = mdai.Client(domain='public.md.ai', access_token="Place Your Token Here")
خروجی کد بالا که نشون میده با موفقیت کلاینت mdai رو نصب کردید:
Successfully authenticated to public.md.ai.
قدم سوم: تعریف پروژه:
با استفاده از id پروژه میتونید یک پروژه در سرئر mdai ایجاد کنید برای گرفتن id کافیه به لینک زیر برید:
https://public.md.ai/annotator/project/{project_id}
برای مثال، project_id میتونه xxxx برای مسیر https://public.md.ai/annotator/project/XXXX
باشه و یک مسیر یا path هم باید بهم بدیم. مثل زیر:
p = mdai_client.project('aGq4k6NW', path='./lesson2-data')
که خروجی اون به صورت زیر میشه:
Using path './lesson2-data' for data.
Preparing annotations export for project aGq4k6NW...
Using cached annotations data for project aGq4k6NW.
Preparing images export for project aGq4k6NW...
Using cached images data for project aGq4k6NW.
قدم چهارم: تنظیم label ids:
در قسمت کد label ids انتخاب شده باید صراحتا توسط توسط روش Project#set_label_ids تنظیم شود تا به نوعی دیتاست آماده شود.
برای اینکار کد زیر را بنویسید:
p.show_label_groups()
که خروجی آن به صورت زیر می شود:
Label Group, Id: G_Vdj4o2, Name: Default group
Labels:
Id: L_A8Jm3d, Name: Lung
سپس کد زیر را بنویسید:
# this maps label ids to class ids as a dict obj
labels_dict = {
'L_A8Jm3d': 1, # Lung
}
print(labels_dict)
p.set_labels_dict(labels_dict)
که خروجی آن به صورت زیر می شود:
{'L_A8Jm3d': 1}
قدم پنجم: ایجاد دیتاست آموزش (Training) و اعتبار سنجی (Validation):
کد زیر را بنویسید:
p.show_datasets()
که خروجی آن:
Datasets:
Id: D_rQLwzo, Name: Dataset
سپس کد زیر را بنویسید:
dataset = p.get_dataset_by_id('D_rQLwzo')
dataset.prepare()
image_ids = dataset.get_image_ids()
len(image_ids)
که خروجی آن:
۱۳۸
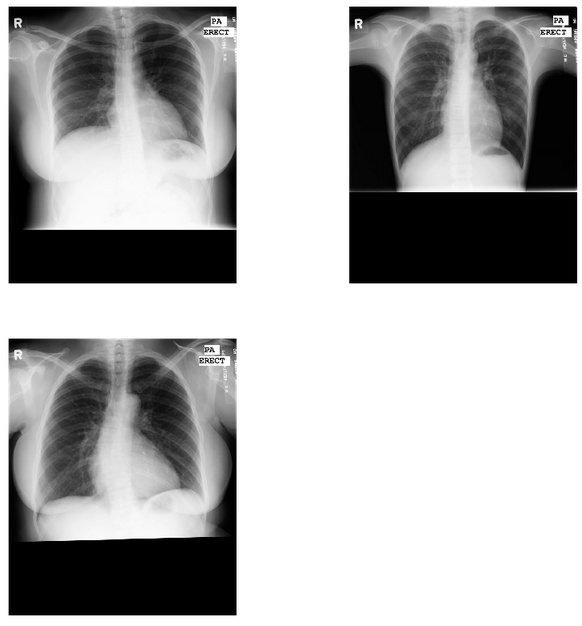
قدم ششم: نمایش چند تصویر:
کد زیر را بنویسید:
# visualize a few train images
mdai.visualize.display_images(image_ids[:3], cols=2)
که خروجی عکسهای زیر خواهد بود:
لینک درس های منتشر شده:
